极大关注!Nature子刊论文一作与读者唇枪舌战16个回合,通讯作者不胜其烦下场开撕……
更新时间:2022-03-25
近日,论文交流平台Pubpeer一篇稿件成功引起小编的注意。这篇刊发在Nature子刊IF高达20+期刊的论文,连翻遭学者提问,作者与读者之间已多轮过招,留言讨论高达16次!讨论内容涉及图片重复使用和原始数据分析等多个方面。

其中一位远在美国的通讯作者因网站给其发送过多提醒邮件而控诉“这种行为太不专业”!

稿件信息:
Gao X, Xia X, Li F, et al. Circular RNA-encoded oncogenic E-cadherin variant promotes glioblastoma tumorigenicity through activation of EGFR–STAT3 signalling[J]. Nature Cell Biology, 2021, 23(3): 278-291.
图源:谷歌学术、西北大学个人主页

图源:中山大学
质疑内容
1、Ext. Fig5图片重复使用:
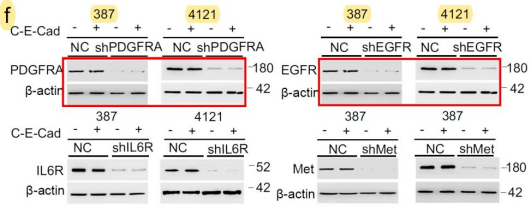
通讯作者张弩回复:
论文修改时间紧迫,非常抱歉没有进行认真的审查。早在交稿时,原始数据已经和稿件一起提交到期刊。
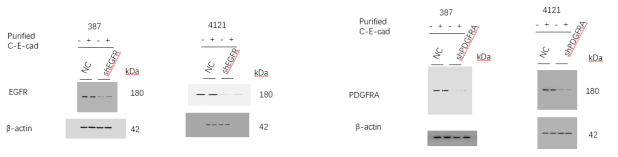
学者再发问:
PDGFRA分子量应该是190kDa?参见: https://www.cellsignal.com/
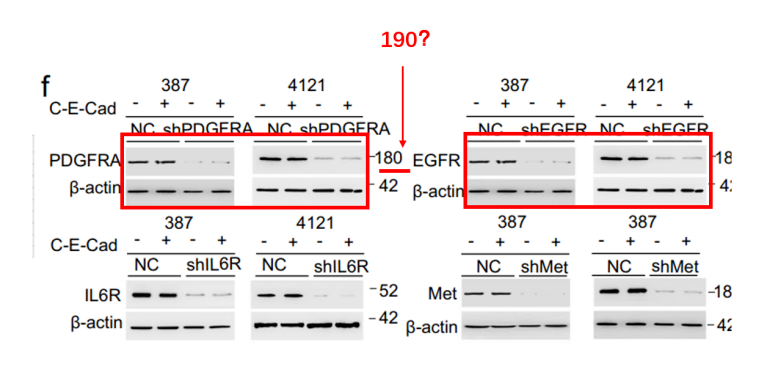
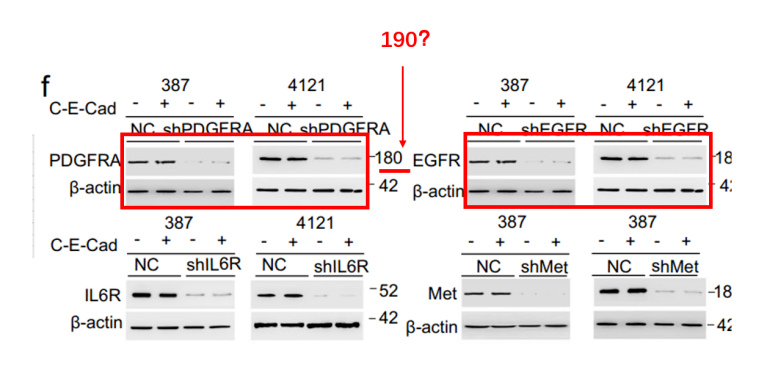
此外,Extended Data Fig. 5图示缺失?

2、Extended Data Figure 6d红色框的两个DNA电泳图几乎相同,作者似乎只是将一幅图的亮度进行了调节,得到了这两张结果。
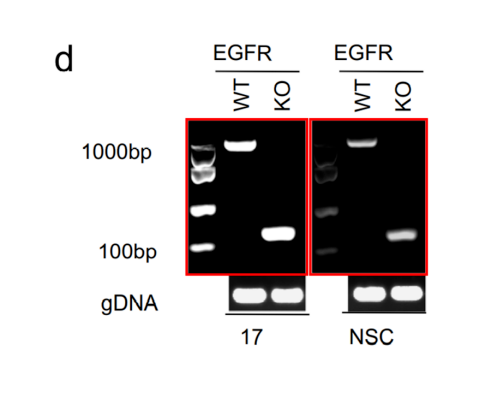
如果将左边图增亮70%,就能清晰地看出这两张图片来源同一个DNA电泳结果。
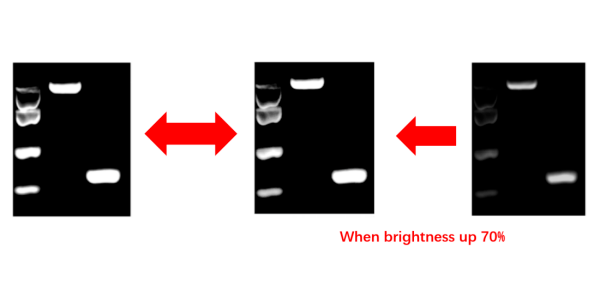
一作Xinya Gao回复:
我们仔细检查了原始数据。我们把GSC17和NSC高曝光和低曝光的照片放在了一个文件夹中,这导致了我们对Ext.Fig.6d的错误粘贴。但是,NSC敲除组细胞的IB和Sanger测序结果能够保证数据完整性,对结论不会有影响。我们将会联系期刊刊发更正。
3、(Litoria Modica提出)Extended Data Figure 2g中间图中病人数量为45?45是cohort 3的数据吧。
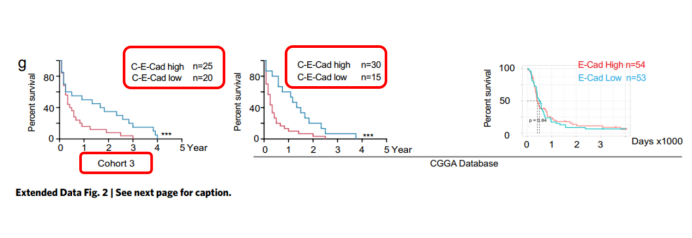
cohort 3高C-E-Cad组与低C-E-Cad组左右图样本量不一致,25 vs 20? 30 vs 15?
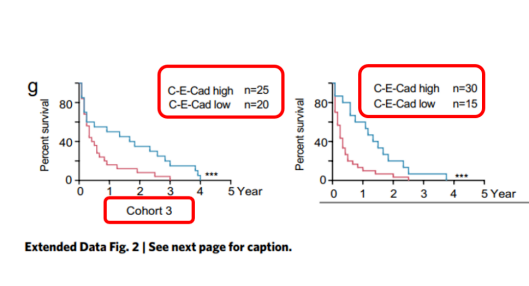
作者回复:
cohort 3和CGGA样本都是独立的群组,两组均是随机选取了45个样本。
学者Pandanus Basedowii再度发问:
感谢回复,我注意到了cohort 3和CGGA样本都是独立的群组。CGGA数据库包含中和右两张图片?图示可有错误?
如你所说,你的数据分析基于CGGA数据,但是,不同阶段,无论是原发还是复发,我都没有重复出同样的结果。您可否上传分析过程?
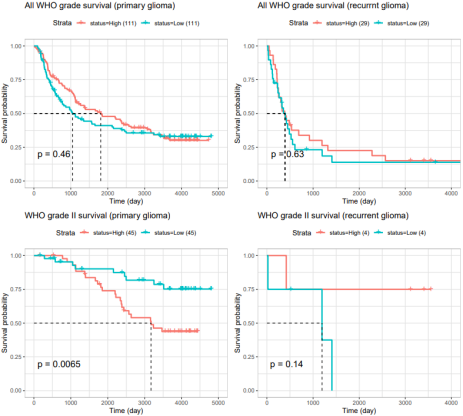

作者能够提供分析所使用的数据吗?
您的论文中Extended Figure 2G似乎并没有使用任何一个数据?E-Cad高表达组样本数为54,E-Cad低表达组样本数为53,合计97。97个样本在CGGA数据库中对应着哪些数据样本?
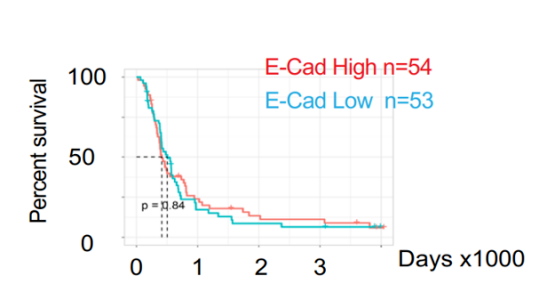
同样,Extended Figure 2G中的107个原始数据是哪些?
到最后,我发现作者使用的是mRNA_array_301数据集、WHO IV glioma。
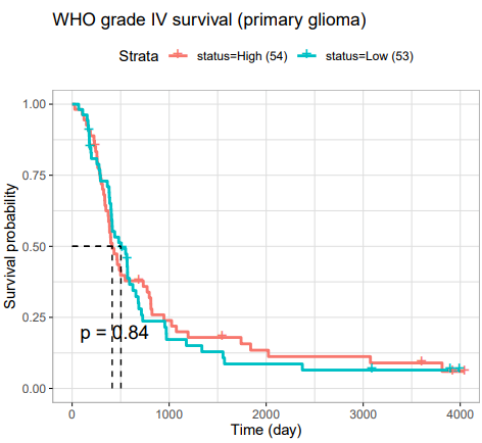
作者为什么不使用mRNAseq_325的结果?mRNAseq_325 dataset数据中E-Cad高表达组与E-Cad低表达组幸存结果更显著P = 0.05。
作者应该解释一下,在CGGA数据库中GBM(比如WHO IV Grade Glioma)mRMA表达微阵列分析结果和mRNA表达测序结果不同的原因。
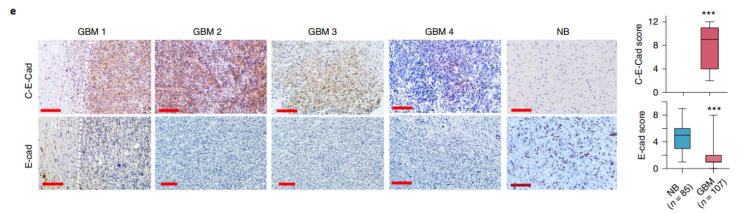
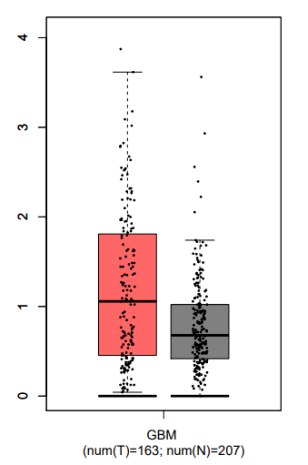
4、作者试图用他们自己的样本来证明E-Cad (CDH1)在正常组织中是高表达的。然而,这个结果与TCGA数据库完全相反。
接下来在Fig. 2F中作者使用TCGA数据库对E-cad在GBM中的生存率分析进行了验证,但是作者有意回避了TCGA数据库中GBM与正常组织E-cad表达的比较结果,只使用了他们自己的样本进行比较。
在TCGA数据库中,E-cad在肿瘤组织中的表达略有上调,但在作者自己的样本中却明显下降,只是为了突出C-E-Cad的作用?
作者回复:
请注意TCGA 数据库并没有包括circular RNA测序结果。正如您所说,肿瘤组织样本中E-Cad RNA表达小幅度上调,但是E-Cad蛋白的表达却是下调的,下调机制是多元的。
5、已上传的原始数据(右图)与论文中,PARP Western Blot条带图片与Caspase3互换。
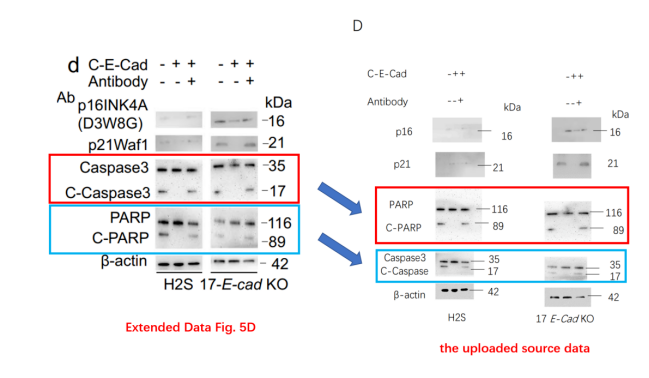
另一位通讯作者Shi-Yuan Cheng就以上问题回应(仅截取部分):
网站一分钟5封邮件,非常不专业!我相信一作已经完全解决了读者的疑问!
此外,实验和生物信息学的相关分析都是张弩在广州完成的,我仅负责对文章整体的重新组织和重新分析大部分数据。如对图片和数据有疑问请找张弩。

这场围绕着图片重复使用、数据合理性的大辩论在PubPeer上如火如荼上演。
目前来看,图片重复使用是作者粗心且无意地拼贴造成的,并不会影响论文结果,已联系出版社刊发更正。部分数据问题,作者回答有理有据,逻辑清晰。
其他图片问题和数据问题还有待作者的正面回复,相信作者能给一个令读者信服的答案。
参考文献:
[1] Gao X, Xia X, Li F, et al. Circular RNA-encoded oncogenic E-cadherin variant promotes glioblastoma tumorigenicity through activation of EGFR–STAT3 signalling[J]. Nature Cell Biology, 2021, 23(3): 278-291.
[2] https://pubpeer.com/publications/F9D4AB94CEF8BE18A7FD8A5CF745D3
洞察学术,追踪动态,欢迎关注AEIC






















.png)


.png)


.png)

.png)